L’expression des gènes est un processus contrôlé dont les dysfonctionnements conduisent à des réponses cellulaires inadaptées, voire délétères. Nous étudions plus précisément les mécanismes post-transcriptionnels impliqués, en questionnant l’importance des étapes de traduction ou de dégradation de l’ARN messager (ARNm) dans ces régulations. Nous nous intéressons aussi aux voies de surveillance de l’ARNm qui permettent le ciblage et la destruction des ARNm aberrants/endommagés selon divers contrôles de qualité liés à l’activité du ribosome.
Utilisant la levure Saccharomyces cerevisiae comme organisme modèle eucaryote, nous participons ainsi à l’exploration de nouveaux champs de recherche par la caractérisation de facteurs, de mécanismes et de nouvelles fonctions a priori très conservés dans le règne végétal ou animal.
1/ Une nouvelle classe d’intéraction ARN-ARN, les intéractions mRNA-mRNA.
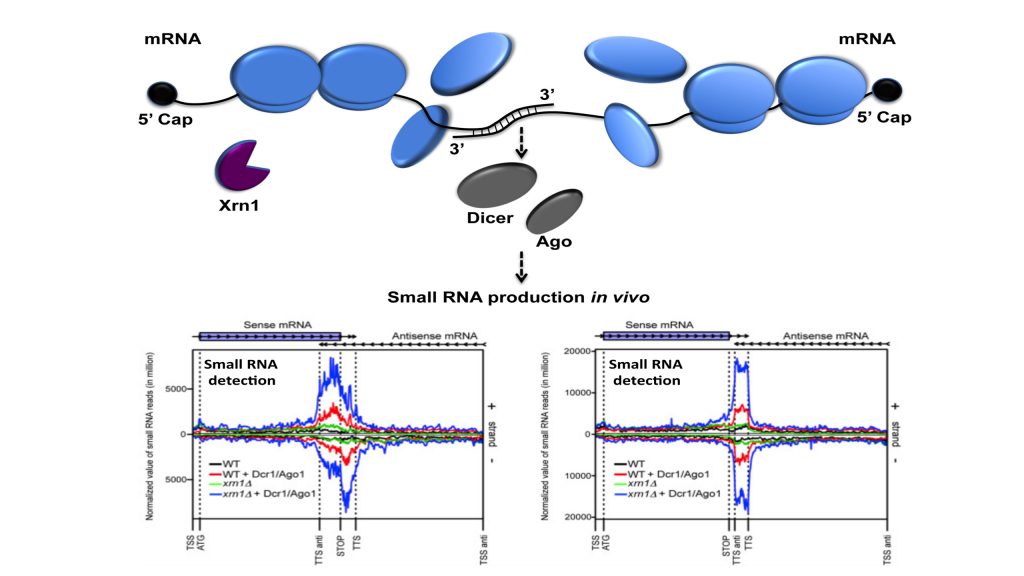
Nous avons montré l’existence d’une large classe d’ARN constituée d’ARNm interagissant entre eux, ARNmim, du fait de la transcription convergente et chevauchante de leurs gènes correspondants. Le point de départ de cette étude concernait l’impact post-transcriptionnel produit par l’inactivation de Xrn1, une exoribonucléase 5’-3’ majeure du cytoplasme et conservée chez tous les eucaryotes. Nous avons observé que l’expression du gène POR1, codant la porine mitochondriale, était particulièrement affectée et causait un défaut respiratoire. Nous avons proposé que l’inactivation de Xrn1 facilitait les interactions entre les régions 3’ des transcrits POR1 et OCA2, et nous avons conclu que la formation de ces séquences, sans doute partiellement ARN double-brin (ARNdb), affectait la traduction de l’ARNm POR1.
Ceci a motivé la caractérisation de ces interactions ARNm-ARNm à l’échelle génomique, en collaboration avec A. Morillon (Institut Curie), aussi bien dans un mutant xrn1 que dans des cellules sauvages. Pour ce faire, nous avons exprimé les protéines Dicer et Ago de S. castelli (un outil développé par David Bartel, Drinnenberg et al., 2009 Science) pour démontrer l’existence de ces duplex ARNm (i.e. ARNdb) in vivo, et révéler l’importance de Xrn1 dans la production de ces interactions à l’origine de séquences ARNdb détectables par Dicer.
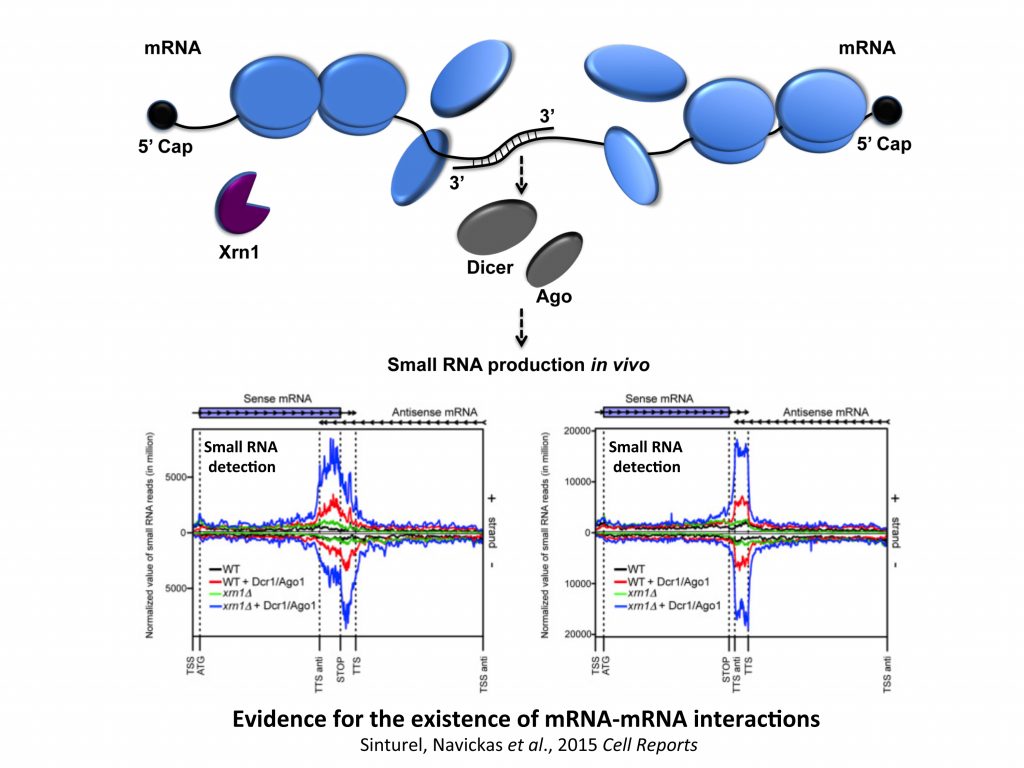
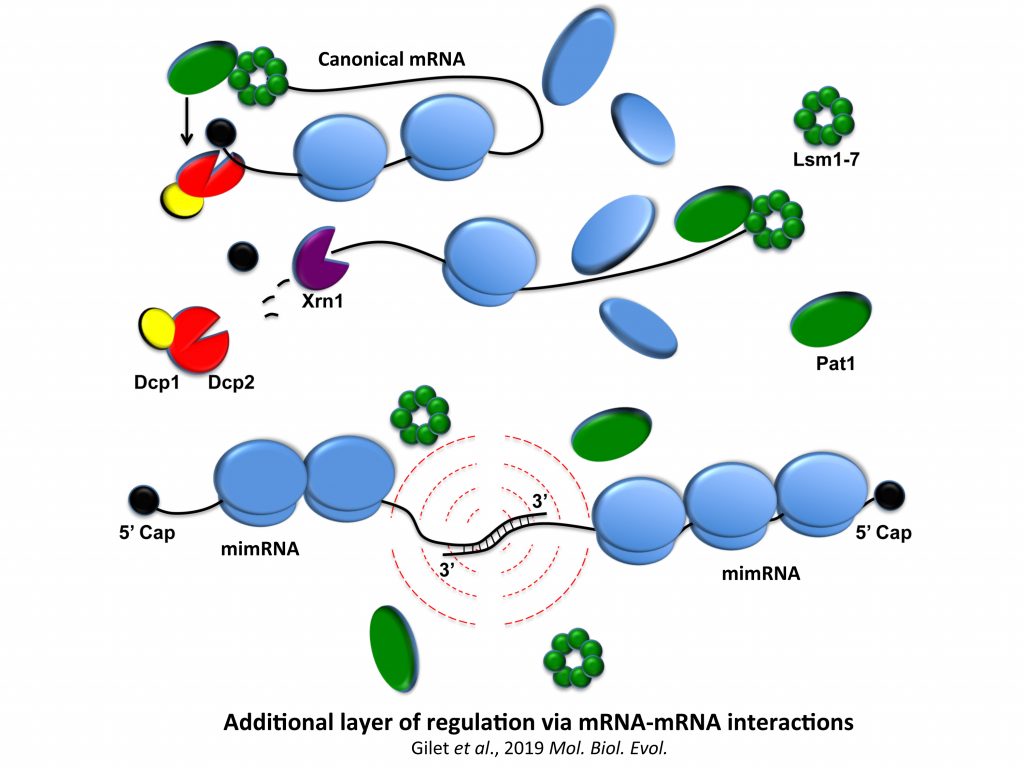
Nos travaux portent aujourd’hui sur l’importance physiologique de ces interactions ARNm-ARNm. Nous avons pour exemple publié un modèle de régulation post-transcriptionnelle médiée par la formation de duplex ARNm selon lequel, lors de stress, le répresseur traductionnel Pat1 lierait préférentiellement les régions 3’ des ARNm canoniques (ceux ne formant pas d’interactions ARN-ARN) et limiterait ainsi l’accès des ribosomes aux régions 5’ des messagers tout en activant l’agrégation de ces messagers (i.e. activant une forme de silencing) au sein de granules. Les duplex d’ARNm échapperaient quant à eux à cette répression en masquant l’accès de Pat1 aux régions 3’ et faciliterait ainsi leur expression continue en condition de stress. Nous proposons aussi, en collaboration avec I. Lafontaine (UMR7141), que ces interactions confèreraient un avantage et auraient été maintenues au cours de l’évolution.

2/ Surveillance de l’ARNm.
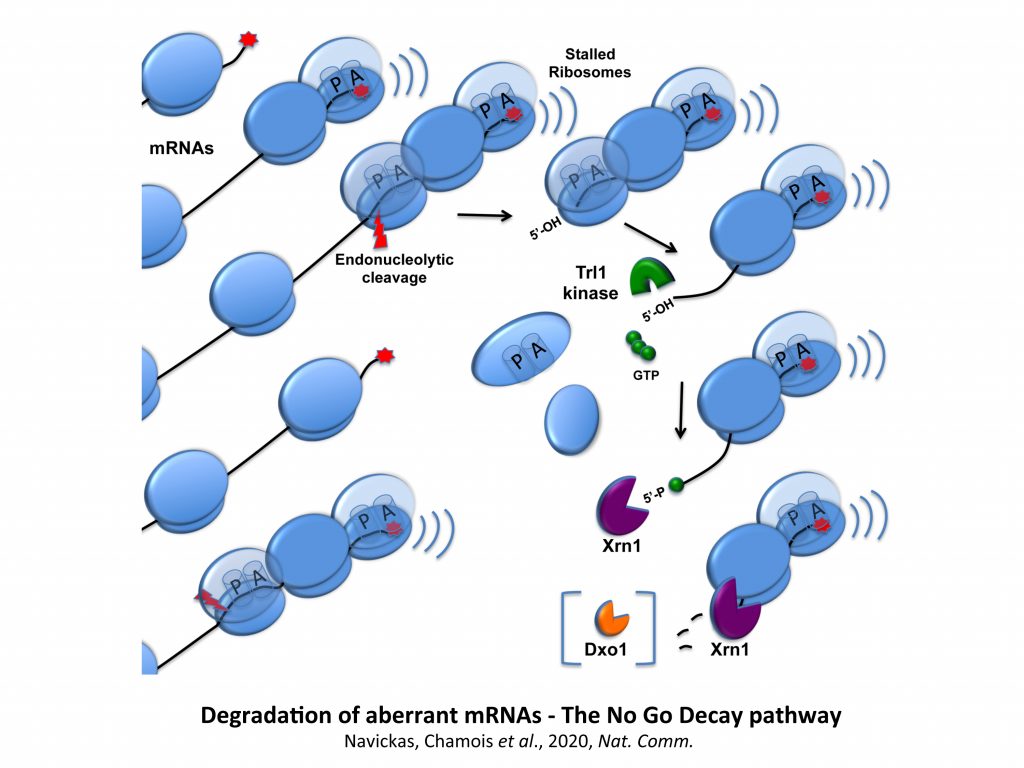
Nous avons montré que des appariements entre ARNm se produisaient au point d’affecter la traduction dans certains contextes et d’activer un évènement post-transcriptionnel particulier comme le No Go Decay (NGD). La voie du NGD est une voie de surveillance de l’ARNm récemment décrite et fait référence ici à la dégradation endonucléolytique et exonucléolytique de messagers pour lesquels l’étape d’élongation de la traduction est fortement compromise.
Ces travaux ont motivé le développement de nouveaux projets portant sur la dégradation des messagers aberrants ou endommagés, cibles de la voie NGD. Pour exemple, nous avons caractérisé avec précision la coupure endonucléolytique activée lors du NGD et démontré que l’activité de la kinase Trl1 était cruciale dans la destruction des fragments ARN produits.

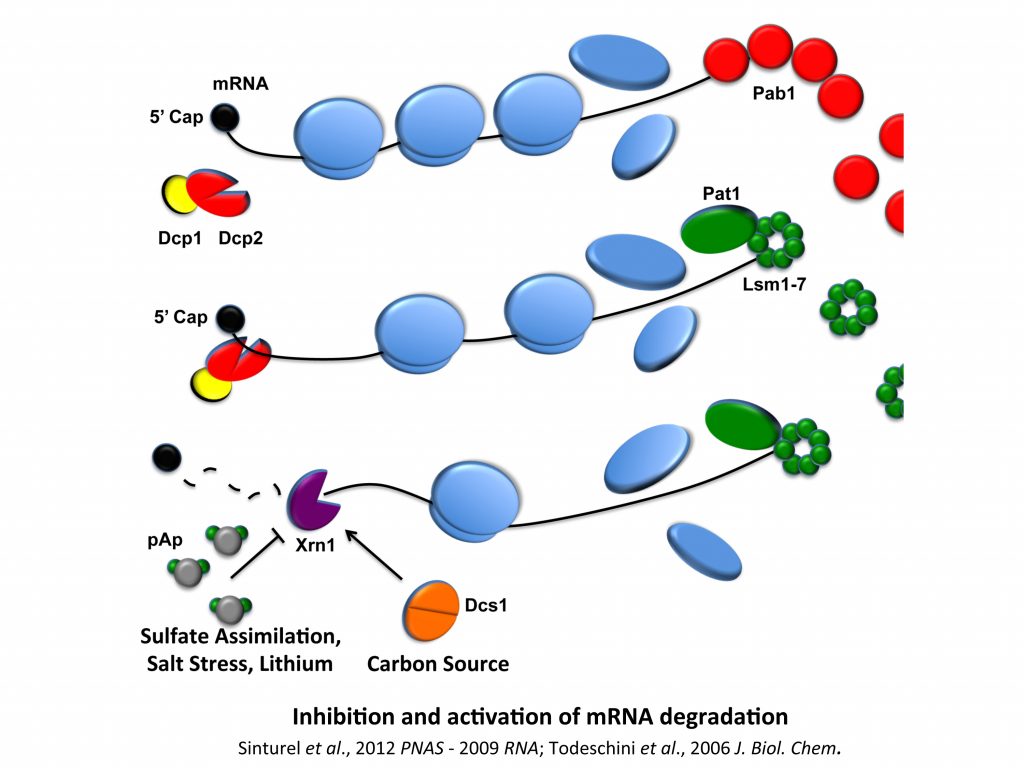
3/ Régulation de l’activité ribonucléasique.
Nous avons un intérêt de longue date pour la régulation des activités ribonucléasiques et de son importance dans la modulation de l’expression génique lorsque, par exemple, la cellule doit faire face à des changements environnementaux. Nous avons par exemple démontré que la cellule limite l’inhibition de l’activité de Xrn1 en contrôlant l’accumulation d’un inhibiteur, le pAp, et que le facteur Dcs1, dont l’expression est corrélée à la nature des sources carbonées, était un co-facteur de Xrn1. Nous avons par ailleurs développé des outils fluorescents pour faciliter la détection ou l’analyse d’activités ribonucléasiques, le développement de ces projets ayant été facilité au sein et en collaboration avec le groupe de C. Condon (UMR8261).

Financements